Pourquoi l’ATTR est un bon “use case” pour l’édition in vivo
L’amylose à transthyrétine (ATTR) est une protéinopathie systémique liée au dépôt d’agrégats de TTR, une protéine tétramérique principalement produite par le foie. Les traitements actuels (stabilisateurs, siRNA/antisens) abaissent la TTR circulante et ralentissent la progression, mais ils ne corrigent pas le défaut génétique et nécessitent généralement des administrations répétées à vie. CRISPR‑Cas9 (ex. NTLA‑2001) a déjà prouvé une forte réduction durable de TTR chez l’humain et est en phase 3, mais le mécanisme DSB + NHEJ peut générer des indels conservant le cadre de lecture (In-Frame Mutations; IFM), un point non trivial quand la protéine cible est intrinsèquement amyloïdogène.
Cas3 en une idée : un système “type I” qui efface plutôt qu’il ne recolle
CRISPR‑Cas3 appartient aux systèmes CRISPR de classe 1 (type I‑E) : la reconnaissance de la cible est assurée par un complexe multi‑sous‑unités (Cascade, chargé d’un crRNA) qui se fixe à l’ADN, forme un R‑loop, puis recrute Cas3. Cas3 est une hélicase‑nucléase processive : une fois activée, elle déroule et dégrade l’ADN de manière unidirectionnelle, conduisant typiquement à des délétions longues (souvent en amont du PAM) plutôt qu’à de petites indels centrées sur une coupure. Pour des indications “knock‑out” (comme l’inactivation de TTR), ce profil peut réduire le risque de conserver une activité résiduelle via des IFM courtes.
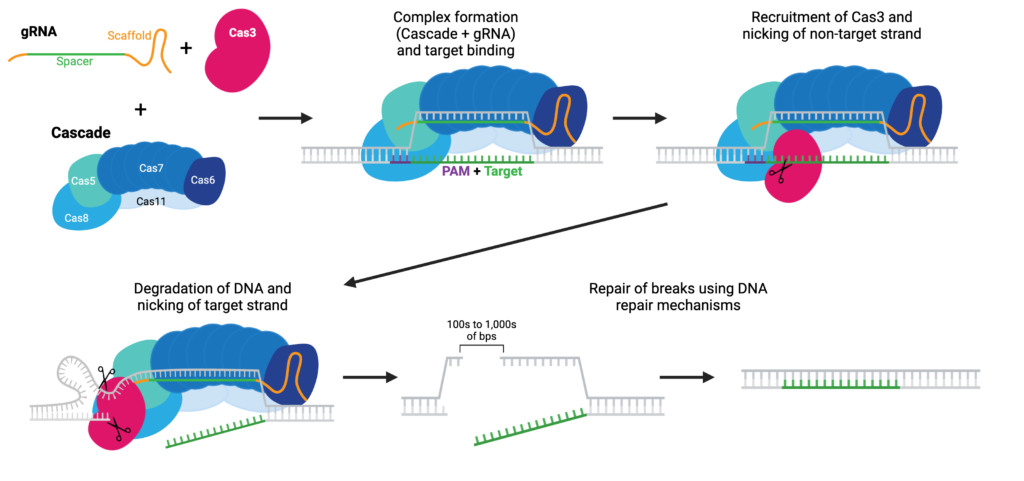
Ce que l’équipe a réellement montré
- In vitro (Hepa1‑6) : après optimisation des crRNA, l’équipe atteint 58,9 ± 0,5% d’édition au locus TTR/Ttr.
- Profil d’édition Cas3 : les délétions sont majoritairement directionnelles et peuvent atteindre ~75 kb in vitro.
- In vivo (foie de souris, LNP) : une injection unique obtient 48,7 ± 1,1% d’édition hépatique et une baisse de la TTR sérique de 80,1 ± 4,6%.
- Taille des délétions in vivo : les délétions observées sont plus “bornées” en conditions LNP, avec une taille maximale d’environ ~21 kb.
- Fenêtre d’expression (cinétique) : Cas3 est détectable dans le foie à 4–6 h, diminue à 24 h, puis devient indétectable à 168 h.
- Transitoire aussi au niveau ARN : les transcrits Cascade sont dégradés en moins d’une semaine, ce qui va dans le sens d’une activité courte et potentiellement plus sûre.
Le “point safety” qui change la discussion : off‑targets et IFM
Sur la spécificité, Cas3 ne montre pas de mutations off‑target reproductibles dans les conditions testées, y compris après WGS short‑read (~139×) et long‑read (~28×), alors que Cas9 présente des événements off‑target détectables à faible fréquence (ex. POT1 et POT129). Mais l’autre différence majeure concerne les produits d’édition : dans le modèle murin à exons Ttr humanisés, Cas3 réduit la TTR humaine sérique d’environ 74,6 ± 0,6% (vs 94,9 ± 0,3% pour Cas9/NTLA‑2001 dans ce set‑up), tout en évitant des IFM détectables dans l’analyse. À l’inverse, dans le groupe Cas9/NTLA‑2001, un peptide TTR mutant (IFM4, p.D39_40TdelinsA) est détecté par nanoLC‑MS/MS, et les auteurs associent cette variante à un comportement plus amyloïdogène dans leurs analyses structurales/biophysiques.
Effets cardiaques + tolérance LNP : les “bonus” de Cas3
Au-delà de la baisse de TTR, Cas3 diminue des marqueurs précoces de pathologie cardiaque dans le modèle exon‑humanized, notamment la deposition de TTR associée à une accumulation de macrophages (signal CD68) observée en immunofluorescence. L’étude décrit en parallèle une réponse cytokine transitoire après injection de LNP (pic précoce puis retour à la ligne de base en ~1 semaine), similaire aux LNP “vides”, ce qui va dans le sens d’une tolérance acceptable à court terme.
À retenir (et à surveiller)
À retenir : Cas3 n’est pas “un Cas9 meilleur”, c’est un éditeur avec un mécanisme différent qui semble particulièrement pertinent quand on veut une inactivation forte d’un gène et minimiser le risque d’IFM résiduelles. À surveiller : le contrôle fin de la taille des délétions, la variabilité inter‑contextes (plasmide vs mRNA‑LNP) et les études de sécurité long terme restent indispensables avant toute translation clinique.
Auteur correspondant : Tomoji Mashimo – Journal : Nature Biotechnology (janvier 2026) – DOI : https://doi.org/10.1038/s41587-025-02949-6